Proteinexpression - Quietly reliable !
Was ist eine Proteinexpression ?
Unser Genosphere BT Gensynthese- und Proteinexpressionsservice (Protein Expression Service) synthetisiert die Gensequenz (100% richtig) und kloniert diese in einen Expressions-Vektor Ihrer Wahl. Durch Codon-Optimierung und Einfügen von „tags“ können wir ihre Sequenz genau auf das ausgewählte Expressions-System abstimmen.

Nicht jedes Protein lässt sich gleich gut exprimieren und anschließend isolieren , daher bieten wir Ihnen verschiedene Expressionssysteme für die Herstellung ihrer Proteine an.
E.coli
Yeast
Insect cells / Baculovirus
Mammalian cells
zellfreies Expressionssystem
Using the right protein expression system for your specific application is critical to success. Consider protein solubility, functionality, purification speed, and yield when choosing an expression system. We offer a wide selection of superior mammalian, insect, yeast, and bacterial protein expression systems to suit your research needs.
Unsere 5 Expressionsysteme
1.) Escherichia coli
The expression of proteins in E. coli is the easiest and quickest approach.
2.) Yeast-Pichia pastoris
First choice for expression of eukaryotic proteins with simple post-translational modifications.
3.) Baculovirus-Insect
Best system for larger MW eukaryotic proteins with post-translational modifications.
4.) Mammalian cells
HEK or CHO cells expression of more complex proteins.
5.) In vitro
Cell-free, well suited for expression of toxic and/or transmembrane proteins.
Die hergestellten Proteine können in einer Vielzahl von Verwendungszwecken genutzt werden, so zum Beispiel zur Generierung von poly- und monoklonalen Antikörpern oder zur Strukturermittlung mittels Kristallographie.
Sequence design
A number of studies have revealed that codon usage bias may cause severe expression problems.
The presence of rarely used codons for the host translation machinery may lead to :
a.) Reduced translation rate of the target mRNA
b.) Amino acids misincorporation,
c.) truncated or amino-acids deleted polypeptides or proteins
d.) Frame-shifted proteins
e.) The overall result being low or undetectable protein production
To help solve the issues related to codon usage bias, the target DNA sequence may be re-defined through codon changes using the appropriate usage frequency table for the host organism: that’s codon optimisation.
This general approach has been verified to significantly improve heterologous protein expression in numerous cases.
Project design process includes adding N-/C- terminus tags for purification and detection purposes.
(*) Protein Expression Service: Failure fee may apply, highly dependent on the actual project and target protein.
Get in contact with our scientific experts
Proteinexpression und Antikörperherstellung
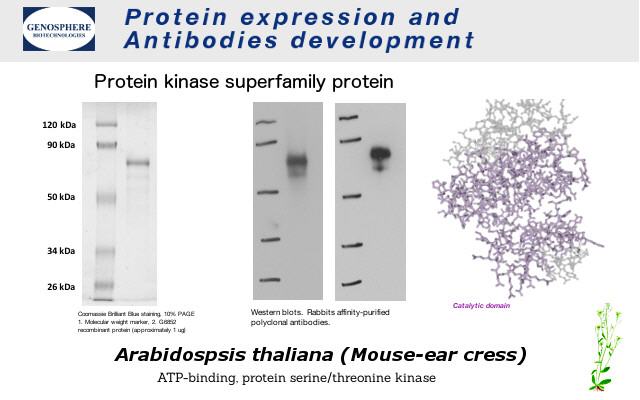
Sie kennen die Gensequenz eines Proteins und wollen einen entsprechenden Antikörper dazu herstellen ?
Unser Genosphere BT Gensynthese- und Proteinexpressionskundenservice synthetisiert die Gensequenz (100% richtig) und kloniert diese in einen Expressions-Vektor Ihrer Wahl. Durch Codon-Optimierung und Einfügen von „tags“ können wir ihre Sequenz genau auf das ausgewählte Expressions-System abstimmen. Wir exprimieren Proteine in prokaryotischen E. coli, im eukaryotischen Baculovirus-Insektenzellen-Expressionssystem, Sf9, sowie in Hefen und Säugetierzellen. Das dabei erhaltene Protein wird gereinigt und zur Immunisierung der Host-Tiere verwendet.
Optional: Tag removal, sofern dies mit guter/ oher Ausbeute durchführbar ist.
Lesen Sie weiter: Von der Proteinexpression bis zum Antikörper, alles aus einer Hand ....
Unsere Site Biomodul im Überblick:
Zugriffe heute: 1 - gesamt: 6754.
